Neste material você vai ter acesso a informações sobre conceitos básicos de marcadores moleculares, tais como:
- O que são marcadores moleculares
- Tipos de marcadores moleculares
- Vantagens dos marcadores moleculares
- Desvantagens dos marcadores moleculares
- Características relevantes de alguns marcadores moleculares
Em vários estudos o uso de marcadores se faz necessário, como uma maneira de acompanhar a evolução de uma espécie ou mesmo por exemplo, para obtenção de linhagens mais adaptadas, produtivas ou resistentes aos patógenos.
Oposto aos marcadores morfológicos (quando se usa uma marca do fenótipo, por exemplo, a cor da pétala) e bioquímicos, os marcadores moleculares não dependem do tipo de tecido analisado ou do estágio de desenvolvimento do indivíduo.
Além de proporcionar várias vantagens em relação aos marcadores fenotípicos tradicionais, uma vez que fornecem dados que podem ser analisados de forma objetiva.
Nas últimas décadas, os marcadores moleculares têm transformado diversos ramos das ciências biológicas, como a taxonomia, ecologia, engenharia genética, genética de populações, genética evolutiva, genética quantitativa e o melhoramento genético de plantas e animais, entre outros.
O que são marcadores moleculares?
Os marcadores moleculares são diferenças hereditárias na sequência de DNA na posição correspondente do cromossomo homólogo de dois indivíduos diferentes, que seguem um padrão de herança mendeliana simples.
Um marcador molecular é um locus polimórfico (com variabilidade na sequência genética) que nos informa sobre o genótipo do indivíduo e sua análise está orientada a todo genoma e é independente das condições do ambiente.
Na atualidade, os marcadores moleculares são usados com as mais diversas finalidades e estão sendo constantemente modificados para aumentar sua utilidade (Quadro 1).
A descoberta da reação em cadeia da polimerase (PCR) foi fundamental na popularização dos marcadores moleculares como ferramenta para a marcação de genes, mapeamento genético, clonagem baseada no mapeamento de genes de interesse agronômico, estudos de diversidade genética, análise filogenética e seleção assistida por marcadores de genótipos desejáveis.
Porém, existem várias técnicas moleculares, diferentes em seus princípios e métodos que devem ser levadas em consideração na escolha do(s) marcador(es) apropriado(s). Estes métodos de marcação molecular podem ser agrupados em duas grandes categorias:
- os marcadores do tipo RFLP (análise de restrição de fragmentos polimórficos) e os baseados na PCR.
Tipos de Marcadores Moleculares
Os principais marcadores baseados em PCR são:
- RAPD (Randomly Amplified Polymorphic DNA)
- AFLP (Amplified Fragment Length Polymorphism)
- SSR (microssatélites ou Simple Sequence Repeat)
- Sequenciamento de fragmentos de DNA
Marcadores do tipo RFLP
Os RAPD’s consistem na amplificação ao acaso de fragmentos de DNA genômico usando um ou mais oligonucleotídeos, que permitem amplificar diversos pontos do genoma.
A RFLP é uma técnica da primeira geração de marcadores, baseada na digestão por enzimas de restrição, de origem bacteriana, das sequências de DNA genômico.
Após a digestão do DNA, é gerado um grande número de fragmentos com distintos tamanhos, que são hibridizados a uma sonda de DNA marcada radiativamente ou por fluorescência química para identificar os fragmentos homólogos num gel de agarose ou poliacrilamida. As mutações nos locais de restrição na região reconhecida pela sonda são responsáveis pelo polimorfismo detectado.
Quando dois oligonucleotídeos hibridizam a distâncias que variam entre 200 e 2000 pb, em cadeias opostas do DNA, a região delimitada entre ambos é amplificada na reação de PCR. O polimorfismo detectado é devido às mutações tanto nas regiões amplificadas, como nos locais de fixação dos primers.
Marcadores do tipo AFLP
Os AFLP’s são uma análise conjunta do polimorfismo nos locais de restrição enzimática e na hibridização de primers não específicos, usando simultaneamente enzimas de restrição para clivagem do DNA e amplificação por PCR dos fragmentos obtidos na digestão.
É essa combinação da enzima de restrição e dos oligonucletídeos que permite a identificação de polimorfismo entre indivíduos.
Marcadores do tipo Microssatélites
Os marcadores microssatélites estão constituídos por sequências repetidas de 2 a 6 nucleotídeos e são encontrados dispersos em todo o genoma.
A análise de variação entre indivíduos é realizada mediante a amplificação por PCR, usando oligonucleotídeos específicos para as regiões que flanqueiam os microssatélites.
É o par de oligonucleotídeos (senso e antissenso) que constitui o marcador. O polimorfismo é representado pela variação no número de elementos repetidos que constituem o microssatélite, produzindo alterações no comprimento dos fragmentos.
Sequenciamento de fragmentos de DNA (SNP’s – Single-Nucleotide Polymorphisms)
Recentemente, os consideráveis avanços do sequenciamento genômico têm permitido determinar as sequências completas do DNA de vários organismos e a possibilidade de compará-las.
Isto, entre outras coisas, permite a identificação de polimorfismos nas sequências devido à deleção/inserção ou mutação de apenas um nucleotídeo.
Estes marcadores se conhecem como SNP’s (Single-Nucleotide Polymorphisms) e são mais abundantes que os microssatélites.
Portanto, aumentam a chance de sucesso em diversas aplicações, como a construção de mapas genéticos de alta resolução, diagnóstico genético, análise da estrutura genética da população, filogenia, entre outras.
| Marcador | Vantagens | Desvantagens |
| RAPD´S | Baratos Produz um grande número de bandas, que podem ser caracterizadas individualmente (Ex: convertidas em marcadores únicos) | Baixa reprodutibilidade Dominantes Difícil de analisar Difícil de automatizar Estudos comparados são difíceis |
| SNP´S | Baixa taxa de mutação Abundantes Fácil de genotipar Novas abordagens metodológicas Facilidade para estudos comparados Banco de dados |
Alta taxa de heterogeneidade entre sítios Viés de apuração Baixo conteúdo de informação em um único SNP |
| Microssatélites |
Altamente informativos (grande número de alelos, heterozigosidade alta) Baixo viés de apuração Fácil de isolar |
Alta taxa de mutação Modelo evolutivo mutacional complexo Difíceis de automatizar Estudos comparados requerem uma preparação especial |
| Isozimas |
Baratos Protocolos universais |
Requer material fresco ou congelado Alguns loci mostram instabilidade da proteína Número limitado de marcadores Alvo potencial de seleção |
| Sequenciamento de DNA |
Maior número de resoluções possíveis Sem viés Estudos comparados são fáceis Bancos de dados disponíveis |
O mais custoso que os outros marcadores |
Quadro 2. Características relevantes de alguns marcadores moleculares importantes na biotecnologia agrícola.
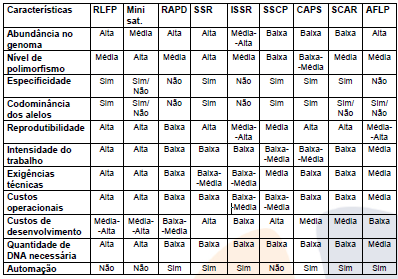
- RFLP: Restriction Fragment Length Polymorphism,
- Mini sat.: minissatélites,
- RAPD: Randomly Amplified Polymorphic,
- SSR: microssatélites ou Simple Sequence Repeat,
- ISSR: Inter Simple Sequence Repeats,
- SSCP: Single-Strand Conformation Polymorphism,
- CAPS: Cleaved Amplified Polymorphic Sequence,
- SCAR: Sequence Characterized Amplified Region,
- AFLP: Amplified Fragment Length Polymorphism.
Referências
Modificado de Kesawat MS & Kumar Das B. 2009. Molecular Markers: It’s Application in Crop Improvement. J. Crop Sci. Biotech. 12(4): 169-181. doi No. 10.1007/s12892-009-0124-6.